The Innovation Life | 森瑞斯最新科研成果发表:用 GPT 模型加速功能性蛋白质发现!
发布时间:
2025-05-30

近日,森瑞斯生物信息科学家曾梓硕博士在The Innovation姊妹期刊 The Innovation Life 发表了Article文章“Accelerating functional protein discovery with GPT models: Antimicrobials and enzymes”。
导 读
你是否想过——蛋白质AI生成式模型可以生成自然界中不存在的蛋白质?然而,如何高效引导AI大模型去生成具有目标功能的序列,以及如何进一步对这些人工序列进行筛选,尚未有系统性研究。
森瑞斯科研团队经过技术攻坚,成功突破AI蛋白质设计瓶颈!通过全球领先的“生成+判别”双模型框架,首次系统性解决了AI蛋白质设计中的微调策略、序列筛选、功能引导等关键难题,对设计新型功能性蛋白质起到事半功倍的效果。
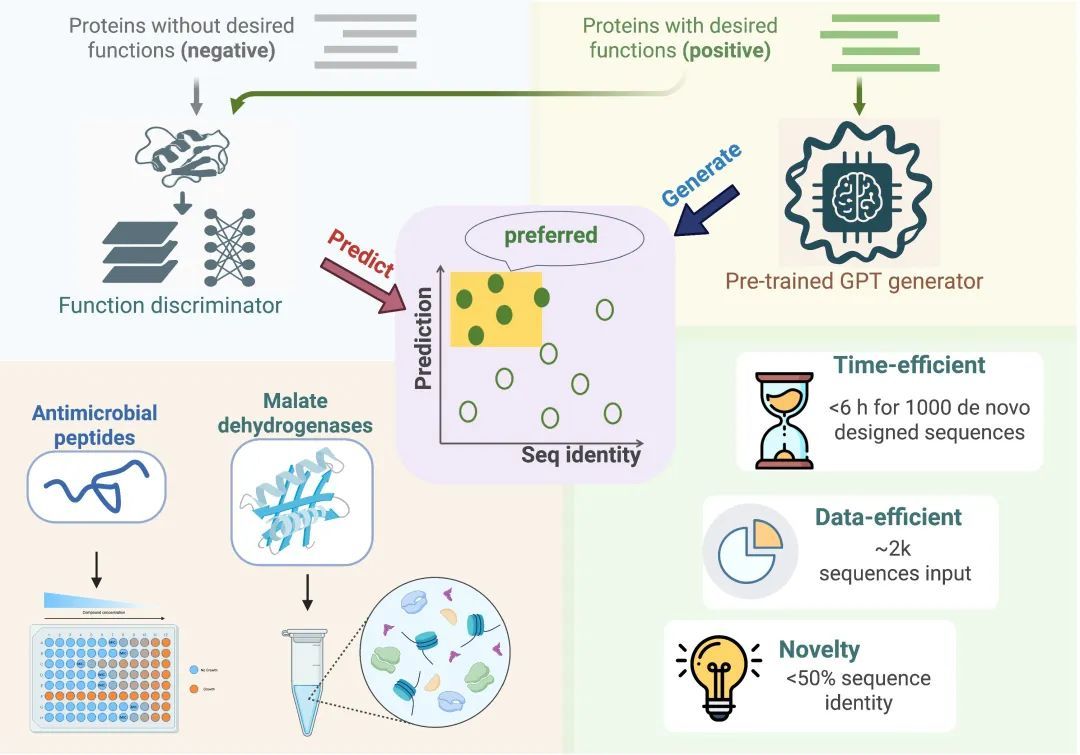
图1 研究概览。森瑞斯科研团队提出“生成模型 + 判别模型”的AI蛋白质设计框架:只需数百条已知序列、6小时计算,就能设计并筛选上千条候选
此前大模型的“打开方式”尚未得到系统研究——主要存在以下几个重要问题:
1. 为了让这些大模型生成人们想要的蛋白质,需要对这些大模型进行微调。但微调序列的数量要达到多少才合适?
2. 微调序列的多样性是否会影响生成结果的新颖性?3. 为大模型提供提示词能否引导模型生成更高质量的结果?
4. 理论上大模型可以生成无数个蛋白质序列,这些序列我们当然不能全盘接收;那么我们该如何进一步筛选?
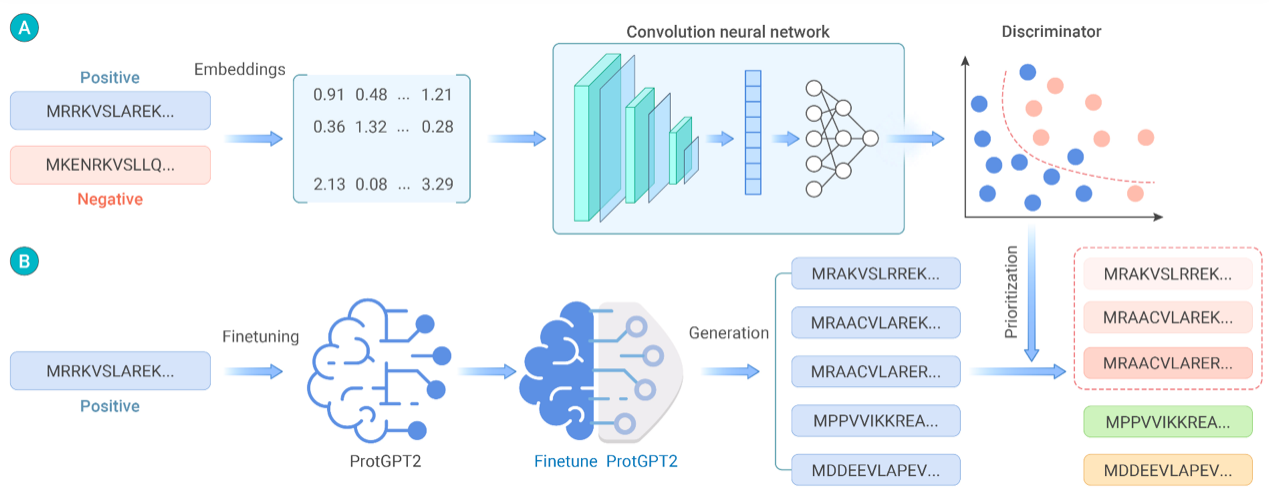
图2 新型蛋白质从头设计框架的搭建——判别模型的构建(A)与生成模型的微调(B)
为了回答这些重要问题,森瑞斯科研团队为一个蛋白质生成式大模型(ProtGPT2)搭配了一个基于卷积神经网络(如图2所示)的判别模型。新框架最关键的优势在于,判别模型可以直接挑选兼顾功能性和新颖性的序列。以抗菌肽和苹果酸脱氢酶这两种功能型蛋白质作为应用案例来测试这套新型蛋白质设计框架,通过计算、实验结果发现:
1、判别模型准确率非常高;
2、微调序列的多样性会直接影响生成结果的多样性;
3、微调序列仅需数百条,大模型即可生成令人满意的结果。同时,引入提示词序列对大模型生成质量的提高并无帮助。
论文链接:https://www.the-innovation.org/article/doi/10.59717/j.xinn-life.2025.100133?sessionid=207546739
总结与展望
这套新型蛋白质设计框架具有数据高效性(仅需数百序列),时间高效性(6小时内生成和筛选1000个候选序列),兼顾功能和创新。
这项突破不仅展现了森瑞斯在合成生物和人工智能交叉领域的深厚积累,更成为行业蛋白质生成大模型的全新“打开方式”!目前,森瑞斯正在积极将该框架用于新型萜类合酶的设计,欢迎广大朋友咨询与合作。
文章来源:TheInnovation创新
相关新闻
2026-01-10
2026-01-10
2026-01-07
2026-01-07
2026-01-04
相关文件
SAF Coolest v1.3.1.2 设置面板 FDJSX-ADJT-ASSVE-ZQQ
无数据提示
Sorry,当前栏目正在更新中,敬请期待!
您可以查看其他栏目或返回 首页



